- 関連リンク
並列コンピューティング技術を使った全ゲノムハプロタイプ関連解析高速計算プログラムparaHaploの開発
2010/06/04
ハプロタイプを基礎とした関連解析ソフトウェアでは、ハプロタイプ推定に時間がかかることがわかったため、ハプロタイプ推定も並列化して高速に行うプログラムも実装しました。これをとりいれたプログラムパッケージを、paraHaplo version 2.0として公開しました。 Misawa K, Kamatani N. 2009. ParaHaplo 2.0: a program package for haplotype-estimation and haplotype-based whole-genome association study using parallel computing. Source Code Biol Med 5:5.
2009/10/21
人間は一人一人の人がそれぞれ自分だけの個性的な遺伝子を持っていて 人によって病気のかかかりやすさやかかりにくさが違います。 このことを逆に利用して、ある病気、たとえば糖尿病や乳がんなどの 病気にかかった人を何千人・何万人と集め、病気にならなかった人を また何千人・何万人とあつめて、その遺伝子を比較すると、 病気にかかった人たちが共通に持つ遺伝子が見えてきます。 このような遺伝子を疾患関連遺伝子と言います。 このような遺伝子は、病気を引き起こす原因である可能性が高く それを調べることで、病気の原因の解明が進み、さらには 病気の治療法や治療薬の開発へとつなげることができます。 このような研究を関連解析と言います。 関連解析により、糖尿病、乳がん、リューマチ、肥満、高血圧など 次々と疾患関連遺伝子が見つかってきています。
遺伝子の一人分のセットをゲノムというのですが、 ヒトのゲノムはDNA分子で30億塩基を両親から受け継いだ 合計60億塩基からなっています。ゲノム全部に関連解析を おこなうことをゲノムワイド関連解析と言いますが、 これは60億塩基×数万人の大量データに関して解析を 行うので、非常に時間がかかります。しかし、患者さん達は 一刻も早い治療を必要としています。
関連解析を高速に行うための、スパコンなどハードウエアの開発と ソフトウエアの開発は、治療薬や治療法の開発を素早く開始するためには 今後は必須となってくると予想されます。
私たちは、SNPがリンクしているという前提での関連解析のType I errorの確率 を、Haplotype頻度から多項分布を利用し、計算する理論式および アルゴリズムを論文を2008年に発表しました。 今年度はこの理論式とアルゴリズムを組みこんだソフトウエアを開発しました。さらに、 並列コンピューティング技術を使い、PCクラスタ上で高速に計算できる ようにしました。 将来のスーパーコンピュータへの移植も視野に入れたアルゴリズムを採用しています。 このプログラムはparaHaplo プロジェクトの成果の一つです。
そのプログラムには、我々のアルゴリズムの他に、 Kimmel and Shamir (2006)のRAT法を使ったハプロタイプ解析のアルゴリズムも組み込みました。 このプログラムはPetaPermutation プロジェクトの成果の一つです。
この両者を含んだプログラムパッケージがparaHaploとしてsourceForgeから公開されています。 http://sourceforge.jp/projects/parallelgwas/releases/?package_id=9706 現時点で、このプログラムを理化学研究所遺伝子多型研究センターに提供し、 47疾患に関する関連解析に利用する作業が始まっています。
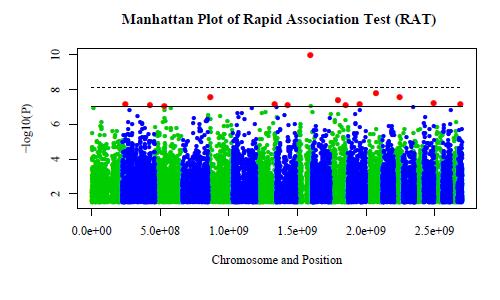
縦軸は患者群とコントロール群の間でハプロタイプ頻度を統計検定した時のP値のlogをとりマイナス1をかけたものです。 この値が高いハプロタイプは疾患関連遺伝子を含むと推測されます。 従来の方法では点線より上に来るものしか検出できませんでしたが、適切な有意水準を定めた今回の方法では実線より上の赤い点をすべて検出できるように なりました。
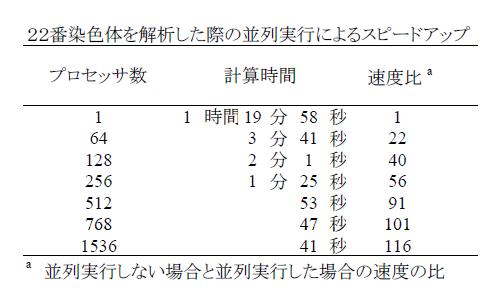
paraHaploはプログラム内部に並列化を実現しているため、 複数の計算ユニットを利用して高速に結果が得られるようになりました。 paraHaploをPCクラスタ上で実行することで、 最大で100倍以上の高速化に成功し、並列化以前は1時間以上かかった解析の結果を1分以下で得ることができました。 しかしこの計算時間を計った際のサンプル人数は200人弱であり、 今後サンプル数が増大すると、PCクラスタでは計算パワーが足りず、 ペタフロップス級のスーパーコンピュータが必要となります。
All-to-all のタンパク質ドッキングとクラスタラリングによるタンパク質間相互作用ネットワーク予測: シグナル伝達系への応用
2009/12/07
背景
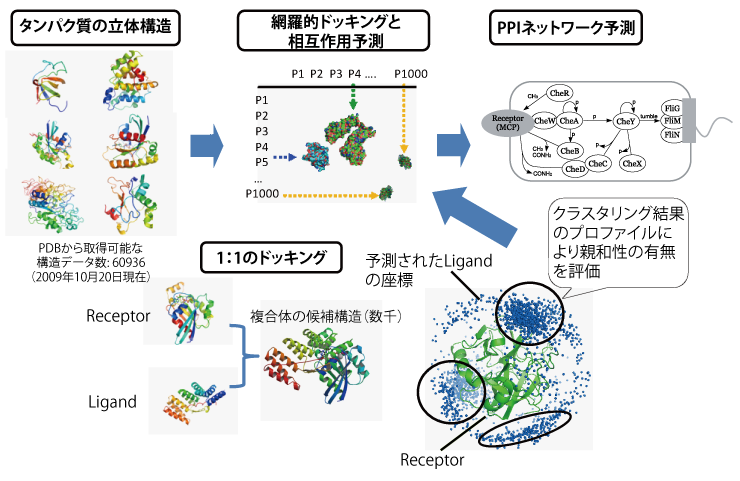
タンパク質間相互作用(PPI)ネットワークの解明は生命の理解における中心的課題の一つである。一方、タンパク質機能の本質的理解には三次元構造の考察が不可欠であり、現在までに6万余件の構造データが公開データベース(PDB)に蓄積されている。しかし、従来のPPIネットワーク予測研究においてはこれらの立体構造データが十分には活用されていなかった。本研究の目的は、形状相補性等に基づく網羅的タンパク質ドッキング計算を大規模に実行し、ドッキング計算結果のプロファイルを解析することにより、ターゲットタンパク質間の相互作用ネットワークを推定することである。
手法
まず、Protein Data Bank(PDB)からタンパク質の構造データを取得して総当たりの網羅的ドッキング計算を行い、出力された数千の結合ポーズ予測結果を結合構造の類似性に基づきクラスタリングした。次にクラスタの密度やドッキングスコアの偏りをもとに各タンパク質対の相互作用可能性の有無を判定し、その集合としてのPPIネットワークを予測した。大規模PPIネットワークの予測を目標とするため、本研究でのドッキングには剛体を仮定 したリジッドドッキングを用いた。
結果
一般的にドッキング性能の評価に用いられるベンチマークデータ(Protein-Protein Docking Benchmark Ver. 2.0)を対象にPPIネットワーク予測を行い、予測モデルの最適なパラメータやクラスタリング手法を決定した。さらに、パスウェイ構造のほとんどが 調べられており、システム生物学の主要なモデル系の一つである細菌の走化性系を対象としてデータセットを構築し、生物学の実問題での評価も行った。この結果、一般的なベンチマークデータで良い精度(F値 0.44)を得たものと矛盾しないパラメータ範囲において、 細菌走化性系の相互作用をランダムな予測より有意な精度(F値 0.49)で予測することに成功した。また、 False-positiveとした相互作用の中でも、実際に相互作用する可能性を実験等で確認する価値のある興味深いタンパク質対(Personal communication, Dr. Sang-Youn Park, Harvard Medical School)について、予測された複合体構造を入力として、さらに他のタンパク質との相互作用予測を試みた。
Reference
Matsuzaki, Y., Matsuzaki, Y., Toshiyuki, S. and Akiyama, Y., In silico screening of protein-protein interactions with all-to-all rigid docking and clustering: an application to pathway analysis. Journal of Bioinformatics and Computational Biology 7(1): 991 – 1012, 2009.
複数SNP間の交互作用を考慮した高速ゲノムワイド関連解析
2009/12/06
国際HapMapプロジェクトの成功などにより,全ゲノム領域をカバーする効率的なタグSNPセットが見出され,今やゲノムワイド関連解析による疾患の原因遺伝子の解明が展開されつつある.これまでは,検出力の問題などにより単一遺伝子座と疾患とのケースコントロール関連解析が多かったが,多因子遺伝病では,疾患が複数の遺伝子によって引き起こされる可能性がある.とくに遺伝子間に相互作用が存在する場合は問題が複雑となり,高度な統計モデルが必要となる.一方,ゲノムワイド関連解析で問題となるのが多重検定の補正である.ボンフェローニ補正は簡便であるが保守的すぎ,ゲノムワイドで数十万のSNPを解析するときに問題となる.これをモンテカルロ法により解決する方法がランダムパーミュテーションテスト(SPT)であるが,計算量が極めて大きいのが難点である.これに対し近年Importance Samplingを用いた高速なパーミュテーションテストであるRapid Association Test (RAT)が提案され,単純なカイ2乗検定による関連解析で良好な成果をあげた.本研究ではRATを拡張し,相互作用を考慮したゲノムワイド関連解析手法を提案する.そのための第一段階として,遺伝子型の組み合わせで疾患に関連する2個のSNPを,ロジスティック回帰で解析する手法を,RATの拡張により実装した.この手法の有効性を検証するため,相互作用つきの2SNPの遺伝モデルに基づき,現実の連鎖不平衡を反映したシミュレーションを行った.その結果、SPTよりもはるかに高速に現実的な時間で,SPTと同等の実用上十分な精度で,ボンフェローニ補正よりも高い検出力が得られることが確認された.本研究の手法は,拡張性が高く統計学的にも確立した手法を組み合わせて実装するため,疾患原因遺伝子間の相互作用がより複雑な場合にも柔軟に適用できるだろう.今後,数万人から数十万人規模のケースコントロール研究がこれから現実化することを考えると,本研究のような2SNPあるいはそれ以上の複数のSNPにもとづき相互作用を考慮した複雑な解析を行うことが必要となる.そしてパーミュテーションの規模も爆発的に増えることから,本研究のような高速化の手法と,次世代計算機の開発との両方があってようやく,疾患により深くアプローチした解析が現実化すると考えられる.(2009年アメリカ人類遺伝学会で発表,論文準備中)
がんゲノムデータ解析による突然変異の網羅的同定
2009/12/06
DNA配列決定技術の進歩により、ヒトゲノムの全配列が1カ月程度で決定できるようになった。しかしながら、得られる情報が短いDNA配列の断片(<100bp)であることや、エラーが多いこと等の問題があり、配列の違い(多型や突然変異)を正確に同定するアルゴリズムは未だに確立されていない。我々は、次世代シークエンサーのデータを用いて、正確に個人間のゲノム配列の違いを同定するために(1)一塩基置換同定、(2)挿入欠失の同定、(3)コピー数多型同定のための手法を開発した。(論文準備中)。
がんの全ゲノムシークエンスを決定することを目指し、国際がんゲノムコンソーシアムが発足した。国際がんゲノムプロジェクトでは、各がん種について、500のがんゲノムと正常ゲノムの配列が決定される予定である。我々は、上記の手法を用いてがんゲノム中の突然変異の網羅的同定を行う予定である。
また、がん細胞は正常細胞に比べて突然変異率が高いため、がんの原因となる突然変異の他にも機能的に中立な突然変異が多く存在することが知られている。我々は、がんの原因となる突然変異と中立な突然変異を区別する手法の開発も行っている。
ヒト全ゲノム上のコピー数多型の大規模網羅的解析
2009/12/06
コピー数多型はヒトゲノム上に普遍的に存在する多様性の一つであり,病気との関連が近年になって注目されつつある.我々は共同研究により,東京大学先端研の油谷研究室が独自に開発・実験を行ったCNV検出の高密度マイクロアレー/チップのデータを網羅的に解析した結果,4千ものコピー数多型を見いだした.それらの多くは,従来の方法では解像度の問題やセグメント重複という難しい領域のために検出できていなかったもので,今回新しく同定されたものである.今回の高分解能の解析からわかったことは,コピー数多型の構造は,大変シンプルで,医学や遺伝学の解析が容易に行える可能性が高いことである.我々は,我々自身が世界に先駆け初めて開発した,コピー数多型とSNPのハプロタイプフェージングを行うアルゴリズムを今回のデータに適用し,コピー数多型のより詳細なハプロタイプ構造を解析することに成功した.その結果,コピー数多型を生むアレルは大変頻度が低いこと,そしてその種類も0,1,2個にほとんど集約されることがわかった.また欧米やアフリカの集団などの違いによって大変異なるコピー数を持つ領域を特定した.そして欠失アレルの頻度が高くなるにつれて周辺のSNPとの連鎖不平衡が強くなるという,コピー数多型の解析上極めて重要な知見を見いだした.その結果,頻度の高いコピー数多型はSNPによって間接的にとらえることができるが,頻度の低いコピー数多型は,医学応用などでは直接観測すべきであるという知見が得られた.このような解析結果は,大規模集団の一人一人の全ゲノムをシークエンスする必要性を示し,次世代シークエンサーからの全ゲノムの網羅的データを解析する上で必須となる次世代スーパーコンピュータの必要性を強く示唆する結果となっている.この研究成果をまとめた論文が2009年12月5日にHuman Molecular Genetics誌からオンラインで出版されました.
Kato, M., Kawaguchi, T., Ishikawa, S., Umeda, T., Nakamichi, R., Shapero, M.H., Jones, K.W., Nakamura, Y., Aburatani, H., and Tsunoda, T. Population-genetic nature of copy number variations in the human genome. Human Molecular Genetics, doi:10.1093/hmg/ddp541 (published online on December 5, 2009).
ヒトゲノム中における平衡選択の探索
2009/12/06
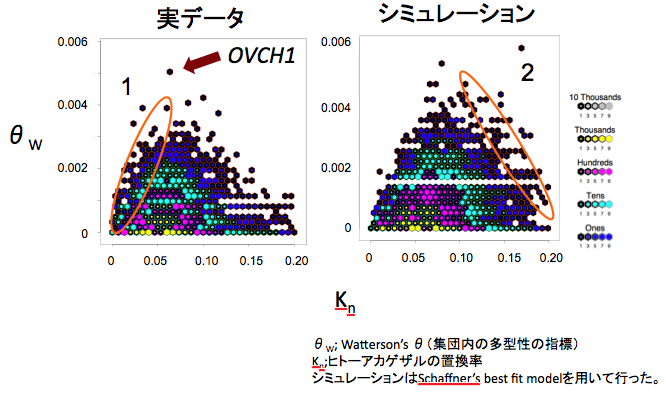
平衡選択とは、複数の対立遺伝子が集団中に長期にわたって維持されることを意味し、正の自然選択と並んで極めて興味深い現象の一つである。しかしながら、平衡選択の検出には、対象とする領域の全変異の検出が必須であるため、平衡選択のゲノムワイドな探索はほとんど行われていない。我々は、平衡選択のゲノムワイドな探索を目的として、JSNPプロジェクトで公開された24人の日本人のエクソン領域とプロモータ領域に関するSNP探索データの解析を行った。平衡選択の影響を受けている領域には、高い種内多型が観察されると考えられるため、ヒトの多型性をヒトとマカク間の塩基多様性で補正を行い、集団構造を仮定したシミュレーションとの比較を行った。その結果、卵の表面で発現しているOVCH1遺伝子において、著しい種内多型が見られることが分かった。また、種内多型が高い遺伝子(上位1%)の遺伝子についてGO解析を行い、機能的偏りを調査したところ、免疫系の遺伝子に加えて代謝系の遺伝子(熱産生や血圧調節)に関連する遺伝子に高い種内多型が観察された。これらの結果より、平衡選択は免疫系の遺伝子に加えて、生殖や代謝に関連する遺伝子においても平衡選択が働いている可能性が示唆された。 この研究成果は、2009年度日本人類遺伝学会および2009年アメリカ人類遺伝学会で発表し、現在論文投稿準備中です。
図: ヒト集団内の多型性とヒトーカニクイザル間の置換率(実測値)とシミュレーション(ヒト24人のゲノムワイドなSNPデータを用いて推定)
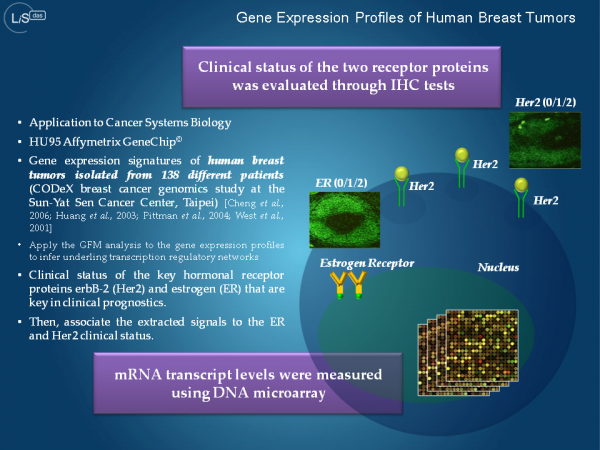
転写制御信号の大規模逆解析と癌研究のためのバイオマーカー探索
転写の最終制御因子は,転写因子と呼ばれるタンパク質の複合体です.転写因子は,遺伝子の上流配列に埋め込まれたプロモータ領域に結合します.このDNA- タンパク質間の相互作用が標的遺伝子の転写開始を制御しています.実際の転写過程は段階的かつ複合的に起こります.コアとなる転写因子がプロモータに結合すると,細胞内に存在するRNAポリメーラーゼと呼ばれる酵素や転写調節因子を呼び寄せ,DNA鎖上で転写因子複合体を形成します.転写因子の活性・不活性 型への変換には,転写因子複合体の組み合わせに応じた選択的なオン・オフ調節機構が主要な役割を担っています.また,転写効率の調節についても,細胞内に存在する転写因子の組み合わせとその濃度変化が大きく関与しています.細胞の分裂や増殖を制御する細胞周期関連因子の生化学反応や体内時計遺伝子が刻む mRNA転写量の概日周期変動は,転写制御ネットワークが本質的な役割を担う典型的な生命現象です.
本研究では,遺伝子発現情報から転写制御ネットワークの制御シグナルを逆解析するための計算アルゴリズム(汎用的アニーリングアルゴリズム)の開発を行いました.生体内の転写制御メカニズムを明らかにするには,転写因子と標的遺伝子からなる因果ダイアグラム(グラフ)を復元することが本質的です.転写制御に関わらず生化学反応系をシステムとして理解するには,ネットワークの全体像を把握する必要があります.しかしながら,生化学反応系ネットワークの構造学習では,NP完全と呼ばれる組み合わせ最適化を解く必要があり,現在の汎用計算機の演算性能では全く歯が立ちません.本研究で開発したアニーリング法は膨大な解空間に無数に存在する不適切な局所解を回避しながら,約3000個の離散変数と同じく約3000個の連続量のモデルパラメータに対して,最適事後分布を安定的に解くことができるアルゴリズムです.本手法を乳癌トランスクリプトーム解析に適用した結果,Her2タンパク質が過剰発現しているヒト乳癌細胞において,17q12と呼ばれる染色体脆弱領域の遺伝子増幅が癌の表現型に関与していることが明らかになりました.本研究で開発された計算アルゴリズムは,次世代スーパーコンピュータGCアプリ「LiSDAS」に実装されます.
中規模遺伝子ネットワーク推定におけるベイジアンネットワークの条件付き最適解を求めるためのアルゴリズムを開発
2009/11/27
ベイジアンネットワークは、遺伝子ネットワークのモデルとして広く用いられ、その有効性は様々な論文で示されている通りです。 真にベイジアンネットワークの性能を発揮するためには、最適なネットワーク構造を求める必要があります。 しかしながら、最適なネットワークを求めるためには、膨大な計算を必要とします。 最大最適ネットワークは、我々の研究グループが求めた31ノードのものが世界記録です。 そこで、大きいサイズのネットワークに対しては、局所最適解を探す学習アルゴリズムが用いられ、11月26日の記事にもありますように全ゲノムを網羅可能とするアルゴリズムが開発されています。
本研究では、ノード数が400以下で、平均次数 (注1) が4以下のようなネットワークであれば、推定されるネットワークの上位構造 (注2) を与えた元で最適なネットワークを推定できる方法 ECOS (Extended Constraint Optimal Search) の開発に成功しました。 また、ECOS により得られる最適解は、Max-min Hill-Climbing などの標準手法として用いられている方法に比べ、2倍から最大10倍程度の推定精度を有することが分かりました。 この方法を用いることで、全ゲノムを対象としたネットワークを NNSR により推定し、より精緻なネットワークを知りたい部分に関して ECOS を用いるという極めて高精度な遺伝子ネットワーク推定戦略を実行することが可能となりました。 この成果により、遺伝子ネットワークを用いた薬剤作用機序の解明や副作用の予測などの応用研究をさらに進めることが期待されます。
本研究の成果は、Journal of Machine Learning Research にて出版予定です:
K. Kojima, E. Perrier, S. Imoto, S. Miyano (2009) Optimal search on clustered structural constraint for learning Bayesian network structure, Journal of Machine Learning Research. (in press)
(注1) 平均次数とは、各ノードに連結するエッジ数の平均です。 (注2) ある無向グラフを設定したとき、推定される有向グラフのスケルトンがその無向グラフの部分グラフとなるとき、その無向グラフを有向グラフの上位構造と呼びます。
ヒト全ゲノムを網羅可能なベイジアンネットワークによる遺伝子ネットワーク推定技術を開発
2009/11/26
ヒト全ゲノムを網羅することが可能な 2 万ノード以上のベイジアンネットワークの推定技術およびソフトウェアの開発に成功しました.ベイジアンネットワークは他の大規模遺伝子ネットワーク推定アルゴリズムと異なり,遺伝子間の発現の依存関係を制御の方向の情報を含めて有向グラフとして推定可能なモデルですが,必要な計算が非常に多いことから大規模な遺伝子ネットワークを高精度に推定することは非常に困難です.
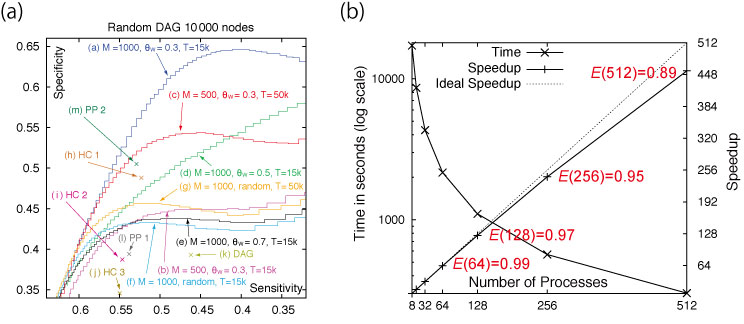
開発した推定アルゴリズム: NNSR (Neighbor Node Sampling & Repeat) アルゴリズムは,このプロジェクトで開発されている大規模遺伝子ネットワーク推定ソフトウェア SiGN の新しい推定アルゴリズムです.本研究では, 開発したソフトウェアを HUVEC ノックダウンマイクロアレイデータ 400 サンプルの約 1 万 3 千遺伝子に適用し、ヒト全ゲノム遺伝子ネットワークの推定に成功しました.開発したソフトウェアは分散メモリ型の超並列型スーパーコンピュータ用に設計され,ヒトゲノム解析センターのスーパーコンピュータの 200 コアを利用し計算を行いました.ソフトウェアは 512 コアまで非常に効率よく並列動作することが確認されており,将来は現在開発中の次世代スーパーコンピュータを用いてより大規模・高精度の遺伝子ネットワーク推定が可能になります.この研究成果は IEEE/ACM Transactions on Computational Biology and Bioinformatics (TCBB) に掲載予定です.
Tamada, Y., Imoto, S., Araki, H., Nagasaki, M., Print, C., Charnock-Jones, D.S., and Miyano, S. Estimating genome-wide gene networks using nonparametric Bayesian network models on massively parallel computers, IEEE/ACM Transactions on Computational Biology and Bioinformatics (in press).
図: (a) 1 万遺伝子のシミュレーションデータを用いた既存の発見的アルゴリズムとの比較.(b) CPU コア数に対するスケーラビリティ.E(n) は並列化効率.
測定誤差を加えた遺伝子ネットワーク推定
2009/11/04
マイクロアレイデータは、測定誤差を伴い計測されます。しかしながら、その測定誤差が どのように遺伝子ネットワーク推定に影響するかは調べられていませんでした。そこで、 本研究では、測定誤差が遺伝子ネットワーク推定に与える影響を実際的な観点から詳細に調べました。 その結果、遺伝子ネットワーク推定に無視できないバイアスを生じさせることを明らかにしました。 そこで、測定誤差の存在を考慮に入れた統計科学的な方法を定義し、新しい遺伝子ネットワーク推定手法 の開発に成功しました。 この成果により、遺伝子ネットワーク推定の精度が格段に向上する状況があると期待され、より高精度な遺伝子 ネットワークを利用したバイオマーカー探索が可能となります。
本研究成果は、BMC Bioinformatics にて発表されます。
Fujita, A., Patriota, A.G., Sato, J.R., Miyano, S. The impact of measurement error in the identification of regulatory networks. BMC Bioinformatics. (in press).
実験条件間のシステム的な違いを網羅的に探索するための遺伝子ネットワーク比較技術を開発
2009/11/03
癌の原因とされる増殖因子や細胞の増殖を抑制する分子標的薬などの外的刺激を加えた場合を想定した複数実験条件下で観測される遺伝子発現データから遺伝子ネットワークを推定し、それにより実験条件間のシステム構造の違い(細胞増殖パスウェイ、細胞分化パスウェイ、薬効パスウェイ、副作用パスウェイなど)を明らかにする遺伝子ネットワーク比較技術を開発しました。
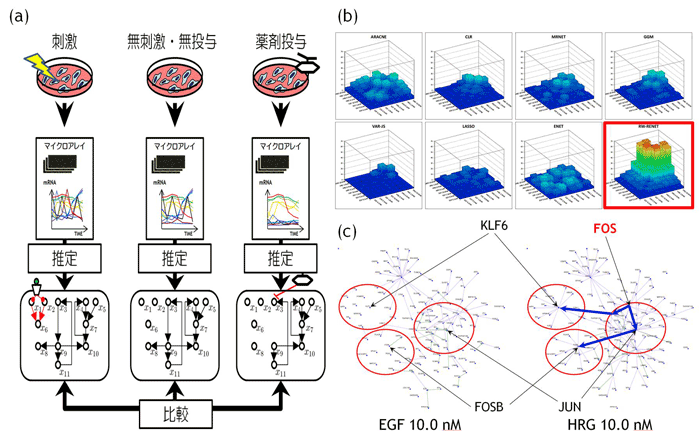
開発したアルゴリズムRelevance-weighted Recursive Elastic NetとアプリケーションソフトウェアNETCOMP(NETwork inference for COMParative analysis)は、従来の研究では想定していなかった遺伝子ネットワーク比較を目的とした初めてのネットワークリバースエンジニアリング技術です。本研究では、開発したアルゴリズムをヒト乳癌細胞に細胞増殖と細胞分化をそれぞれ誘導する増殖因子EGF、HRGを加えた時系列マイクロアレイデータに適用しました。その結果、細胞分化を誘導するHGFにしかみられない複数のハブ遺伝子を抽出し、これらハブ遺伝子がFOSと呼ばれる遺伝子によって制御されている可能性が示唆されました。現在、ネットワーク推定・ネットワーク比較にかかる計算量が膨大なため、100程度の遺伝子ネットワーク比較しか行えませんが、将来的に次世代スーパーコンピュータに実装されることにより、全遺伝子をターゲットにした網羅的な遺伝子ネットワーク比較が可能になる予定です。この研究成果は17th Annual International Conference of Intelligent Systems for Molecular Biology (ISMB) & 8 th European Conference on Computational Biology (ECCB)で発表し、Bioinformatics に掲載予定です。
図:(a) 開発した手法の概念図、(b) シミュレーション結果(赤枠が開発したアルゴリズムによる性能)。これまでのネットワークリバースエンジニア技術よりも高精度にネットワーク推定・比較を行うことが可能。 ©NETCOMPアルゴリズムにより推定された細胞増殖を誘導するEGF刺激(10.0nM濃度)を加えた場合の遺伝子ネットワーク(左図)と細胞分化を誘導するHGF刺激(10.0nM濃度)を加えた場合の遺伝子ネットワーク(右図)。
Shimamura, T., Imoto, S., Yamaguchi, R., Nagasaki, M., and Miyano, S.. Inferring dynamic gene networks under varying conditions for transcriptomic network comparison (in press).
マイクロアレイデータの測定誤差と品質チェック
2009/08/19
マイクロアレイデータは、測定誤差を伴い計測されると言うことは周知の事実です。 さらに、マイクロアレイ間で相関係数が大きいものは、データの再現性も高いと言われています。 しかしながら、相関係数とデータの再現性は当然ながら異なるものです。 そこで、相関係数が大きくてもデータにバイアスが含まれる場合、当然ながら再現性が高いとは言えません。 従って、マイクロアレイデータに含まれる測定誤差を推定する手法を開発し、マイクロアレイの実験の品質を計算により評価する方法を構築することに成功しました。 この研究成果により、遺伝ネットワーク推定に用いるマイクロアレイデータの品質のチェックを事前に行い、ネットワークの推定精度を向上させることが期待されます。 また、マイクロアレイデータ取得のための再実験の必要性も示唆することが可能となりました。
Fujita, A., Sato, J.R., da Silva, F.H.L., Galvao, M.C., Sogayar, M.C., Miyano, S. Quality control and reproducibility in DNA microarray experiments. Genome Informatics. (in press)
非時系列マイクロアレイデータを用いた遺伝子間制御の向きを推定する手法
2009/08/17
時系列マイクロアレイデータに基づく遺伝子ネットワーク推定では、時間の情報を使いるため、 制御関係の向きをデータから推定することが可能です。しかしながら、時系列ではないマイクロアレイデータからは、特殊な状況を除き、遺伝子ノックダウンなどの介入操作を併用しつつ制御関係の向きを調べることが必要になります。 遺伝子間の制御関係の向きは、薬剤作用機序を知るためには極めて重要な情報です。 それらを時系列データだけからではなく、時系列ではない、たとえば多数のがん細胞におけるマイクロアレイデータ などから推定することができれば、非常に多くのデータを利用でき、推定結果も非常に高精度であることが期待できます。
そこで、時系列ではないマイクロアレイデータから、制御関係の向きを調べる contagion と呼ばれる概念を利用し、 遺伝子ネットワーク推定に初めて用いた。 その枠組みのもと、より精緻にパラメータを推定する新しい推定手法や、推定された関係が統計的に有意か否かを判定する ための統計的仮説検定の開発にも成功しました。
本研究は以下の論文で発表されます。
Fujita, A., Sato, J.R., Demasi, M.A.A., Yamaguchi, R., Shimamura, T., Ferreira, C.E., Sogayar, M.C., Miyano, S. Inferring contagion in regulatory networks. IEEE/ACM Transactions on Computational Biology and Bioinformatics. (in press).
世界最大の最適ベイジアンネットワークの推定に成功
2009/8/11
ベイジアンネットワークはバイオインフォマティクス分野において遺伝子ネットワークのモデルとしてよく利用されるグラフィカルモデルの一つで,マイクロアレイデータなどの観測データから遺伝子間の発現の依存(因果)関係を予測するために用いられます.しかし観測データに最適なベイジアンネットワークの構造を探索することは非常に計算量の多い問題として知られており,特にメモリ量の制限が,ノード数の大きいネットワークの構造を探索する際のボトルネックになっています.これまで報告されているもので実際にデータから構造探索を行ったベイジアンネットワークは計算が高速な離散モデルを用いたものでノード数29が最大でした.
当研究分野では過去に動的プログラミング法による最適ネットワーク構造推定アルゴリズムを世界で最初に発表しております (Ott et al. 2004).このアルゴリズムは動的プログラミング法を用いて非常に効率よく最適ネットワークの構造推定を行うことが出来ますが,共有メモリ型の計算機で実行されることが前提であるため,現在標準的になっているスーパーコンピュータの型式である分散メモリ型の超並列型計算機では実行することができません.共有メモリ型のスーパーコンピュータはメモリ搭載量及び計算速度に限界があり,現在理化学研究所が中心となって開発を進めている次世代スーパーコンピュータも分散メモリ型のスーパーコンピュータです.
今回,当研究プロジェクトでは動的プログラミング法による最適ネットワーク構造推定アルゴリズムに基づく新しい解空間の分割法を発明し,分散メモリ型計算機で実行可能な効率の良い実装によるプログラムの開発に成功しました.このプログラムをノード数30,サンプル数50のシミュレーションデータに適用し,ヒトゲノム解析センターのスーパーコンピュータの256コアを使用し計算させたところ,約86時間後に正常に終了し,世界最大サイズであるノード数30での連続値モデルを用いた最適ベイジアンネットワークの構造推定に成功しました.使用したメモリは総計で約255.5GB(各コアあたり約1GB)でした.この研究成果は現在論文投稿準備中です.
薬剤の作用機序解明のための動的ベイジアンネットワーク解析
2009/7/24
Araki, H., Tamada, Y., Imoto, S., Dunmore, B., Sanders, D., Humphrey, S., Nagasaki, M., Doi, A., Nakanishi, Y., Yasuda, K., Tomiyasu, Y., Tashiro, K., Print, C., Charnock-Jones, DS., Kuhara, S., Miyano, S. Analysis of PPARalpha-dependent and PPARalpha-independent transcript regulation following fenofibrate treatment of human endothelial cells. Angiogenesis, 12(3), 221-229, 2009.
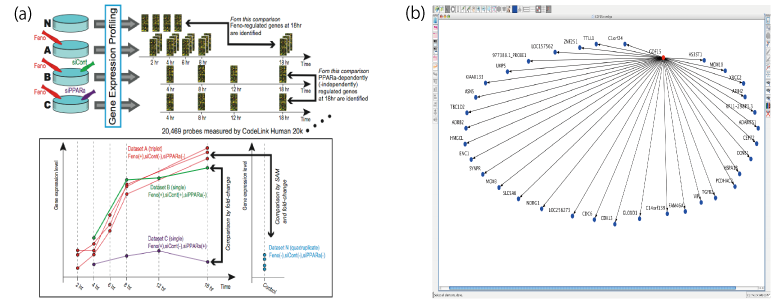
薬剤の作用機序解明は副作用の予測や、薬剤のレスポンダー、ノンレスポンダー選択時に用いるバイオマーカーの探索などにおいて極めて重要である。本研究では、血管内皮細胞での抗炎症・抗増殖作用が明らかになっていない Fenofibrate を投与した HUVEC の時系列データから時点毎に変動遺伝子群を抽出し、このプロジェクトで開発されている大規模遺伝子ネットワーク推定ソフトウェア SiGN を用いて、動的ベイジアンネットワークを推定した。推定された遺伝子ネットワークと既知の標的遺伝子である PPARα の siRNA を投与した実験結果から、Fenofibrate は HUVEC では PPARαを介さない経路を制御して、抗炎症・抗増殖作用の働きを行なっている可能性が示唆され、それらを制御する新規の Fenofibrate 標的候補遺伝子として GDF15 を同定した。
図:(a) 開発した手法の概念図、(b) GDF15(赤丸)を起点とする遺伝子ネットワークの一部。
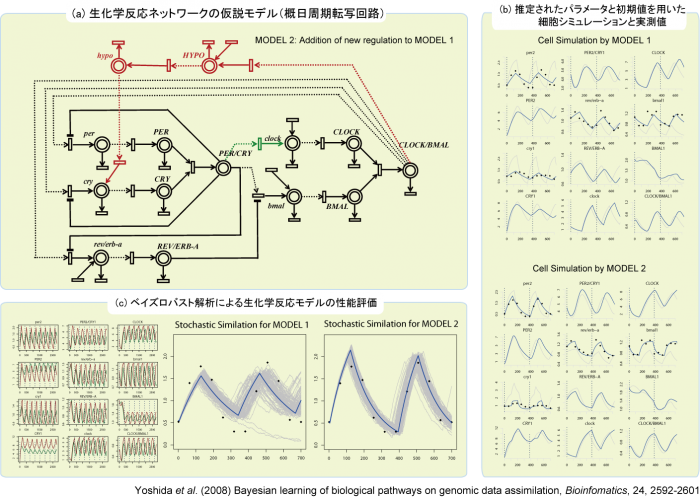
生化学反応系のネットワークモデリングと大規模ベイズ逆問題の数値解法
2009/06/10
常微分方程式で記述された生化学反応ネットワークと反応濃度の時系列データを利用して、未知の分子間相互作用を検出するためのデータ同化技術をBioinformatics誌において発表した。(1) 反応モデルのパラメータ推定(大規模ベイズ最適化問題の数値解法)、(2) システムのロバスト解析にもとづくベイズ型モデル評価、 (3) シミュレーションと観測データの不一致を解消するための仮説モデルの自動生成など、生化学反応系のイン・シリコモデリングと実験生物学を統合するための新しい「統計的イン・シリコ解析」の基礎理論が本論文において提案された。
図:( a ) 生化学的知見を取り込んで作成した生化学反応系の仮説ネットワーク、( b ) ベイズ逆問題を解いて求めたパラメータと初期濃度を用いた数値シミュレーション、( c ) ノイズを混入させシミュレーションに擬似的な揺らぎを生み出し,観測データへの適合度を計測することでモデルのロバストネスを評価する。
GPUクラスタに関するQ & A
2009/12/8 gpuクラスタに関するq_a
Q1. GPUあるいはGPGPUコンピューティングとはなんですか GPUとは画像演算処理装置のことで、PCに内蔵、または周辺機器として接続して使用 します。GPUが搭載する多数の演算器を一般の数値計算に転用して高速化をはかるの が、GPGPUコンピューティング (General Purpose = 汎用)です。また、GPUクラスタ とは、比較的廉価なPCに高性能GPUを接続したものを単位として構成したPCクラスタ のことです。
Q2. GPUクラスタはCPUのみのPCクラスタより廉価なのでしょうか。 A2. はい。確かに、GPUカード一枚で、1TFLOPS/100万円ですから、CPUのみで 同FLOPSのPCクラスタを構成するより廉価だといえます。
Q3. では、今後の高性能並列計算はGPUクラスタに移行してゆくのでしょうか A3. それほど単純ではありません。装置だけを考えるとGPUは非常に廉価な のですが、開発効率、つまり人件費を考えると、逆にGPUの方が高くつきがちです。 これには、GPUアーキテクチャの問題と、ソフトウェア資産の問題等が関係します。
Q4. GPUアーキテクチャの問題とは A4. GPUはあくまでも画像演算に最適なように設計されています。数十のスレッド (並行的な計算の流れ) が同じ計算をしなければならないこと、メモリアクセスが非 常に低速なこと、という制約があります。問題毎に、これらの制約を踏まえたアルゴ リズムを設計する必要があり、人的コストは高くなりがちです。
Q5. ソフトウェア資産の観点はどうでしょう A5. GPU用の専用プログラミング言語 (CUDAなど) で記述する必要があります。 Fortran, OpenMP, MPIなどの言語で記述された資産をそのまま使うことはでき ません。また、CPUではあたりまえに使えたデバッガがGPUでは使えない点も、 GPUでの開発の人的コストを上げる要因となるでしょう。
生命体データ同化と統計的イン・シリコ解析に関するレビュー論文
2009/06/10
吉田亮, 樋口知之, 細胞内生化学反応経路のグラフィカルモデリングと統計的推測手法の新展開. 日本統計学会誌, 38(2), 213-236, 2009.
生化学物質の量的データを利用して、生化学反応ネットワークの数理モデルをいかに構築・再構築していくかという課題は、システム生物学における重要不可避な問題である。ここでいう「ネットワーク」とは、細胞内外で起こる様々な化学反応の連鎖を表す多義的な用語である。具体例としては、転写制御(mRNA の合成)に関わる一連の生化学反応群、タンパク質間相互作用、代謝反応ネットワークなどが挙げられる。モデル開発の過程は概ね次のプロトコルから成る: (1) 文献やデータベースの情報にもとづく仮説モデルの作成、(2) 計測データ(例えば、遺伝子発現データ)にもとづくモデルパラメータの推定、(3) 推定モデルの性能評価、 (4) リモデリング。 本論文では、生化学反応ネットワークのイン・シリコ解析確立へ向けたシステム生物学およびバイオインフォマティクス研究の世界的動向について紹介すると同時に、ここ数年に渡って推進してきたわれわれの研究の成果および現状について報告を行った。また、研究領域に課された諸問題に対して統計科学的観点から整理を行い、予想される今後の研究動向について展望を述べた。
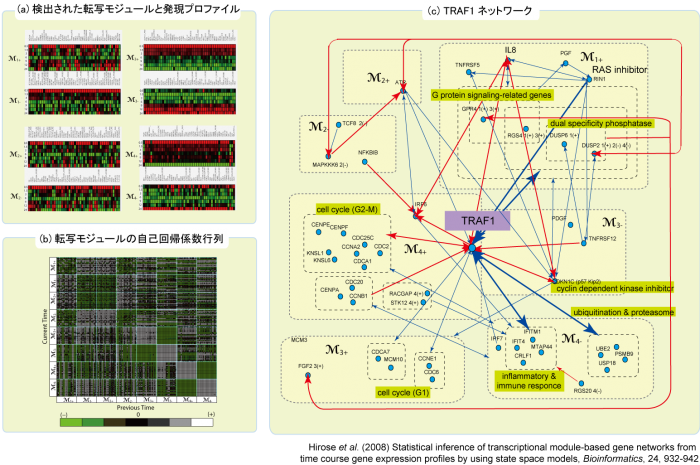
状態空間モデルにもとづく転写制御モジュールダイナミクスのゲノムスケール解析
2009/06/10
転写制御の中心機構である転写因子ネットワークの動的特性を解析するための技術提案を行った。遺伝子発現量の時系列データを利用して転写因子と標的遺伝子群の制御ネットワークを同定することを目的とした,統計的モデリングおよび解析方法を考案した。このデータ解析技術は、観測された発現パターンから数千から数万遺伝子から成る転写制御モジュールとそのダイナミクスを推測するために用いられる。
図:( a ) 1048個のHUVEC関連遺伝子の発現プロファイルから同定された8種類の転写モジュール、( b ) 転写モジュール遺伝子間の相互作用の強度を示す自己回帰係数行列、( c ) HUVECの薬剤応答におけるTRAF1ネットワーク。
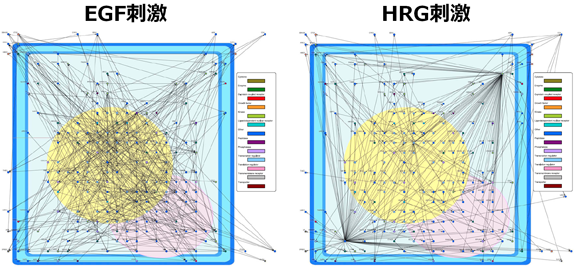
再帰的正則化ベクトル自己回帰モデルによる時系列マイクロアレイデータからの大規模遺伝子ネットワークの推定
2009/04/22
時系列に観測されるマイクロアレイデータから、 数千から数万遺伝子を対象した大規模遺伝子ネットワークを推定する技術として、 再帰的正則化法(Recursive elastic netアルゴリズム)と呼ばれる新しい統計的推定法に基づく ベクトル自己回帰モデルを用いて、遺伝子間相互作用を表すネットワークを推定する方法を開発しました。 この方法を,ヒト乳癌細胞に2つの異なる刺激(EGF刺激とHRG刺激) を加えた時系列マイクロアレイデータに適用することにより、 それぞれの刺激を加えた場合に見られる遺伝子間相互作用ネットワークを再構築し、特に HRG刺激を加えたときにだけ見られるハブ遺伝子を抽出することに成功しました。 本研究で用いられている方法はソフトウェア開発コード:L1GNEに実装されます。
左図:Recursive elastic netアルゴリズムにより推定されたEGF刺激を加えた場合の遺伝子間相互作用を表す遺伝子ネットワーク(ノード:遺伝子、辺:相互作用)。右図:HRG刺激を加えた場合の遺伝子ネットワーク。異なる刺激により、制御されている遺伝子間相互作用が異なることがわかる。特に、HRG刺激を加えた場合にだけ、多数の遺伝子を制御する(多数の遺伝子とリンクする)ハブ遺伝子の存在が確認できる。
CpG突然変異のアミノ酸置換への影響の評価
2009/02/01
DNA上に起こる突然変異は、病気の原因にもなるが、進化の原動力にもなる。 そのため、ヒトの突然変異率の測定は、医学・生物学的に重要である。 ゲノム上でCとGが並んでいるときに、Cがメチル化され、さらに脱アミノ化することで、 非常に高い頻度で突然変異が生じることが知られている。これをCpG突然変異という。 ヒト、チンパンジー、マウスの3種の間で、7645遺伝子、2,954,492コドンを比較することによって、 突然変異のうちどれくらいがCpG突然変異によるものか測定した。 その結果、アミノ酸を変化させるもの、させないもの、どちらについても 全体の14%ほどがCpG突然変異によるものであることが分かった。
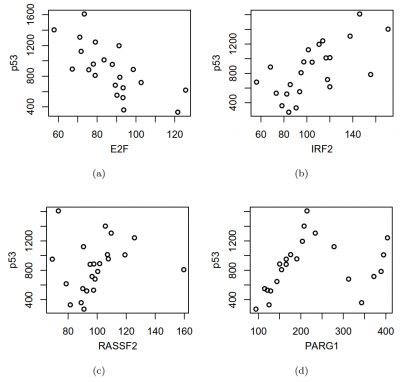
非時系列マイクロアレイデータにおける非線形な遺伝子間制御の研究
2009/01/17
時系列データの場合、ベクトル自己回帰モデルなどの時系列解析手法により遺伝子間の制御を抽出することができます。 また、非時系列データの場合、線形な遺伝子間制御は Pearson の相関係数を調べることにより発見可能です。 非時系列データから非線形な遺伝子間制御を抽出する場合、Mutual Information (MI) が用いられます。 しかしながら、MI においては、その有意性を評価する統計的仮説検定は確立されていないため、その解釈は困難です。 そこで、MI に代わり Hoeffding's D measure を使うことで遺伝子間の非線形な制御を同定する手法を確立しました。
図:(d)のように実際の遺伝子発現データにおいて、非線形な遺伝子間制御も存在していることが分かります
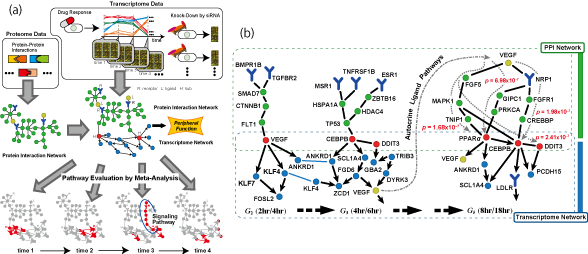
大規模遺伝子ネットワーク推定技術を応用した自己分泌シグナル伝達パスウェイの解析
2009/01/06
自己分泌シグナル伝達パスウェイを制御しているネットワークの解析に成功し、その論文が発表されます。本研究は、まず薬剤応答時系列データから時点毎に抽出した変動遺伝子群に対し、このプロジェクトで開発されている大規模遺伝子ネットワーク推定ソフトウェア SiGN を用いて、それぞれの遺伝子群の遺伝子ネットワークを推定します。そこに既存データベースから構築したタンパク質間相互作用ネットワークを組み合わせることにより、時系列毎に変化するシグナル伝達パスウェイを抽出する新しい手法を提案するものです。この手法を Fenofibrate を投与した HUVEC 時系列マイクロアレイデータに適用することにより、この薬剤に応答した自己分泌シグナル伝達パスウェイを抽出することに成功しました。本論文は 2009 年 1 月 5 日から開催される Pacific Symposium on Biocomuting 2009 で発表されます。
図:(a) 開発した手法の概念図、(b) 抽出したパスウェイおよび遺伝子ネットワーク。上部が PPI ネットワークより抽出した有意なパスウェイ、下部がそれにつながるハブ遺伝子およびその周辺の遺伝子ネットワーク。
ペタスケールコンピューティングに向けた超多粒子フィルタの実装
2009/01/06
Nakamura, K., Yoshida, R., Nagasaki, M., Miyano, S., Higuchi, T. Parameter estimation of in silico biological pathways with particle filtering towards a petascale computing. Pacific Symposium on Biocomputing. 14: 227-238, 2009. [BSCS2008ポスター発表]
データ同化手法の一種である粒子フィルタは並列性の高い手法である.その特性を生かし,超多数のサンプルを用いて遺伝子ネットワークモデルのパラメータ推定を行った.その結果,従来の知見よりも遥かに多くのパラメータについて,妥当な推定が可能であることを実験的に示した.同時に,アルゴリズムの検討と簡易的な時間見積もりを行い,ペタスケールコンピューティングにおいて,妥当な計算時間で結果が得られることを確認した.これはソフトウェアPFに実装されます.
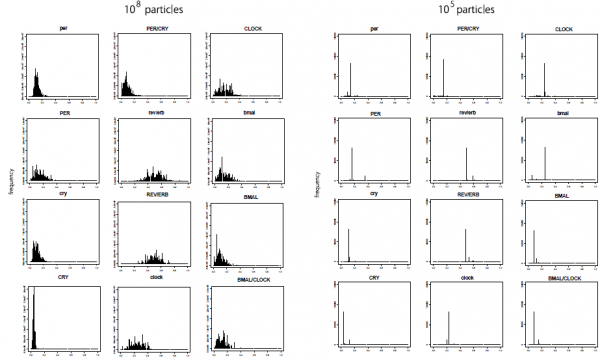 図:10万粒子(右)と1億粒子(左)のそれぞれの場合の初期状態推定の結果.1億粒子の方は適切に滑らかに変化しており,適切な分布の推定が得られているが,10万粒子では滑らかに変化せず,分布としての評価に問題があることがわかる.
図:10万粒子(右)と1億粒子(左)のそれぞれの場合の初期状態推定の結果.1億粒子の方は適切に滑らかに変化しており,適切な分布の推定が得られているが,10万粒子では滑らかに変化せず,分布としての評価に問題があることがわかる.
遺伝子間制御の変化による癌細胞
2008/12/05
正常な細胞と前立腺癌細胞を比べる時、通常は両方の状態での遺伝子発現差 を調べる。だが、発現差だけではなく、遺伝子間の制御にも変化がある事を この研究では解明された。 本研究は以下の論文で発表されている。
図:数万遺伝子の高次元マイクロアレイをデータ圧縮し、正常細胞と癌細胞の判別に関わるバイオマーカー探索を行う。バイオマーカーと見られた遺伝子をネットワーク解析で調べるとその遺伝子の制御関係が変わっていた事が明らかになった。
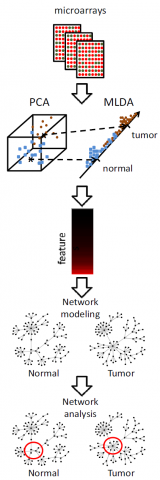
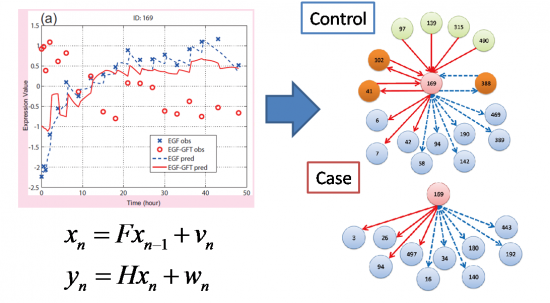
状態空間モデルによる遺伝子制御関係の差異の探索
2008/12/02
Yamaguchi R, Imoto S, Yamauchi M, Nagasaki M, Yoshida R, Shimamura T, Hatanaka Y, Ueno K, Higuchi T, Gotoh N, Miyano S. Predicting differences in gene regulatory systems by state space models. Genome Informatics, 21, 101-113, 2008. [GIW2008口頭発表]
時系列マイクロアレイデータから,統計的時系列モデルである状態空間モデルを用いて, 異なる細胞株間や,薬剤投与条件に差のある細胞間の 遺伝子制御システムの差異に関わる遺伝子群を, モデルからの予測と,観測結果の差異から抽出する新しい手法を構築しました. この手法を,ヒト小気道上皮細胞にEGFを投与した場合と,EGFおよびGefitinibを 投与した場合の時系列マイクロアレイデータに適用することにより, 薬剤投与により制御関係に変異が生じた遺伝子群を抽出することに成功しました. 状態空間モデルによる,遺伝子発現時系列の予測モデル構築には, ソフトウェアSSM が使用されています. 本論文は,2008年12月1日から開催されたInternational Conference on Genome Informatics (GIW2008)で 発表されました.
図:(左)Controlの時系列遺伝子発現データを用いて構築した状態空間モデルから,Caseにおける遺伝子発現パターンの予測を行い,予測(赤線)と観測(赤丸)が大きく異なる遺伝子群を探索し,発現制御関係に差異のある候補遺伝子群を抽出する. (右)候補遺伝子群の制御構造の差異を遺伝子間ネットワークにより推定する.
アミノ酸の増減のUniversal trendはCpG突然変異によって引き起こされる
2008/06/23
2005年に、Jordanらは、多くの生き物で遺伝子配列中のアミノ酸組成の増減に共通した傾向がある ことを見つけ、これをUniversal trendと名付けた。これに対し、Universal trend はMP法のbias による見かけ上の傾向だという批判が多くなされた。まず、我々はMP法のbiasを理論的に解析し、 Universal trendがbiasとは無関係であることを示した。さらに、我々は、Universal trendの原因は CpG hypermutabilityであると結論付けた。CpG hypermutabilityとは、CとGのdinucleotideのC がDNMT酵素によりメチル化され、さらに脱アミノ化することによりTへと化学変化してしまう突然 変異であり、通常の突然変異に比べると数倍から数十倍の突然変異率を持つ。我々の結論は次の2つ の証拠から支持される。(1)CとGのdinucleotideを含むcodonはそうでないcodonよりも減少傾向に ある。(2)DNMTを持たない生物のアミノ酸増減はUniversal trendに従わない。
全ゲノム関連解析の多重検定を補正する方法の開発
2008/06/03
全ゲノム関連解析とは、関連解析とは、患者群とコントロール群でDNA配列情報を比較し、 疾患遺伝子を解明する方法である。具体的には、患者群とコントロール群で 有意に頻度の差があるハプロタイプは、疾患を引き起こしていると判断する。 ヒト一人一人のゲノム上のDNA配列の違いは数十万から数百万にも及ぶため、 患者群とコントロール群でDNA配列情報を比較する時は、多重検定の問題が生じる。 つまり、多数のテストを繰り返すことで、疾患に関係ない場所が 偶然疾患に関係あると判定されることがある。 これをさけるためには個々のテストの有意水準を下げる必要がある。 このような場合に有意水準を際に広く使われている方法はBonferroni補正と呼ばれる方法だが、 これはサイト間に連鎖がある場合は厳しすぎることが知られていた。 そこで我々は、連鎖があることを考慮に入れた上で、多項分布を利用し、 直接確率を計算することで、有意水準を決定する方法を開発した。
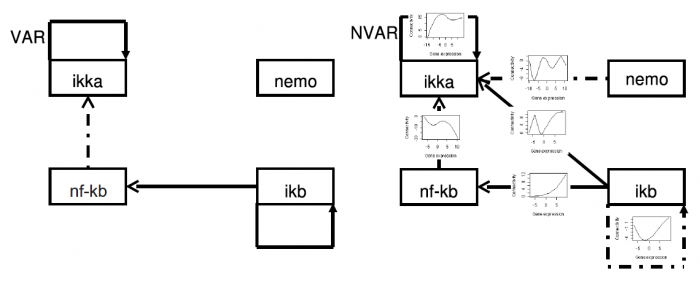
非線形ベクトル自己回帰モデルによる遺伝子ネットワークの推定
2008/02/27
ミカエリス・メンテン式や Hill Function などにみられるように、細胞内の物質の関係は非線形であることが知られている。 それに伴い、遺伝子間制御も非線形でもあると言う事が指摘されている。 しかしながら、これまでの時系列マイクロアレイデータを解析し、遺伝子ネットワークを推定するための自己回帰モデルに基づく方法は、遺伝子間の線形性を仮定した、線形自己回帰モデルに基づくものであった。 そこで、非線形自己回帰モデルに基づく因果関係の発見のために、非線形グレンジャー因果律を定式化し、その推定手法として、新しい非線形ベクトル自己回帰モデル (Nonlinear Vector Autoregressive model - NVAR) の開発に成功した。この方法によって初めて非線形の因果関係が抽出可能となり、遺伝子ネットワーク推定が抽出できる遺伝子間制御の種類が大幅に増した。
図:左が線形ベクトル自己回帰モデル(VAR)、右が非線形ベクトル自己回帰モデル(NVAR)で推定された遺伝子ネットワーク
本研究は、以下の論文で発表されている。
L1正則化グラフィカルガウシアンモデルによる大規模遺伝子ネットワークの推定
2007/12/04
数千から数万遺伝子を対象した大規模遺伝子ネットワークを推定する技術として、 新しいL1正則化法(Weighted Lassoアルゴリズム)を用いたグラフィカルガウシアンモデルによる推定手法を開発しました。 数値実験では、従来法よりも高精度に遺伝子ネットワークを推定可能であることを示しました。 また、この手法をarabidopsis thalianaのマイクロアレイデータに適用し、 イソプレノイド生合成経路として知られるMEP経路とMVA経路のクロストークを抽出することに成功しました。 本研究で用いられているアルゴリズムはソフトウェア開発コード:L1GNEに実装されます。 本論文は、2007年12月3日から開催された International Conference on Genome Informatics (GIW2007)で発表されました。
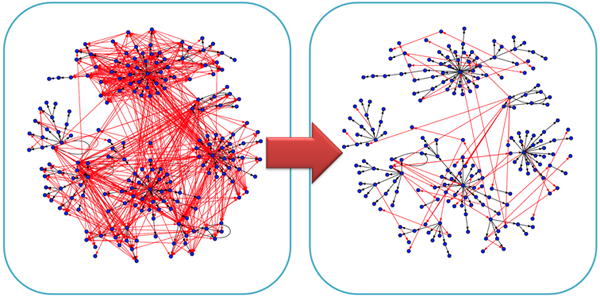
左図:従来法により推定される遺伝子ネットワーク(ノード:遺伝子、辺:相互作用)と右図:Weighted lasso アルゴリズムにより推定される遺伝子ネットワーク。従来法(左図)と比較し、開発した方法(右図)では、実際には存在しないが誤って抽出される擬陽性の遺伝子間相互作用(赤)を大幅に削減し、真に存在する遺伝子間相互作用(黒)を抽出することができる。
スパースベクトル自己回帰モデルによる遺伝子ネットワークの推定
2007/08/30
数百遺伝子を対象した大規模遺伝子ネットワークを推定する技術として、L1 正則化法(Lassoアルゴリズム)を用いたスパースベクトル自己回帰モデル (Sparse Vector Autoregressive model - SVAR) を開発しました。数値実験で は、従来法よりも高精度に遺伝子ネットワークを推定可能であることを示し ました。 また、この手法をHeLa癌細胞のマイクロアレイデータに適用し、 cell cycle関係の遺伝子ネットワークを抽出することに成功しました。
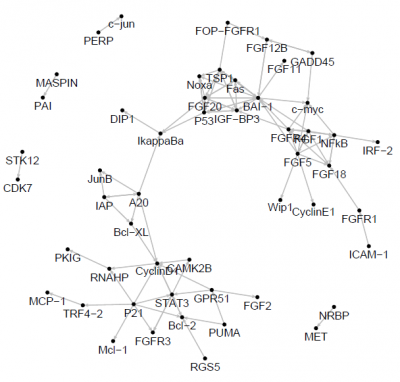
図:SVARを用いて推定されたHeLa細胞のcell cycle遺伝子ネットワーク
本研究は
で発表されました。