【大規模遺伝子ネットワーク推定とその応用】
目的と意義
ヒト全遺伝子産物を対象にした遺伝子ネットワーク推定が可能となり、創薬ターゲット遺伝子のイン・シリコ探索が大規模にできる。宮野等は、産学連携プロジェクトとして、ヒト血管内皮細胞の遺伝子ノックダウンに基づいたマイクロアレイ発現解析を270の遺伝子について行った。この解析データと高脂血症薬(fenofibrate)応答遺伝子群をマイクロアレイ解析で推定することで、べイジアンネットワークとノンパラメトリック回帰を組合わせた独自の方法により、世界で始めてトランスクリプトームネットワークとも呼ぶべき約1000の遺伝子からなる大規模遺伝子ネットワークの構築に成功している。このネットワークを独自に開発した高度可視化ネットワークツールで解析することにより、fenofibrateのターゲット遺伝子であるPPARαの関与するパスウェイを推定し、それから新たな創薬ターゲットの候補を発見している。このネットワーク推定と解析には、200GFLOPSコンピュータシステムを2週間稼動して計算したが、ネットワークとして探索できた遺伝子は約1000が限界であり、これは数としては、ヒト遺伝子数の3%程度である。さらに、生物は、遺伝子ごとの発現制御をしているのではなく、遺伝子に平均5個以上あるプロモーターごとに遺伝子を使い分けているという事実を考えると、少なくとも数万ノードのネットワーク推定とその解析ができなければ網羅的な探索を行ったとはいいがたい。我々の推定アルゴリズムの理論的解析によると、3万個の遺伝子(産物)について、ネットワークを推定しようとすると、2PFLOPSの性能が必要となる。こうしたネットワークの計算が実現すれば、ネットワークを用いて、被影響遺伝子の同定、作用点の推定、副作用の予測・回避、創薬ターゲット・毒性関与パスウェイ探索などが大規模に行える。
ソフトウェアの機能
- 正則化法によるモデル推定
- 大規模な生命分子のネットワークを推定・探索することが可能なプログラム
| 入力 | トランスクリプトーム、プロテオームデータをはじめとする生命システムの観測データ |
|---|---|
| 出力 | 大規模な生命分子のネットワーク構造とダイナミクス |
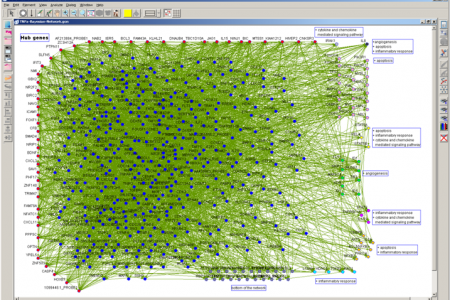
(図1) 351種の遺伝子の siRNA ノックダウンによるDNAチップ解析データから推定されたヒト血管内皮細胞遺伝子ネットワークと同定されたハブ遺伝子群
何ができるか
- 大規模な生命分子のネットワーク構造を「地図」として、シミュレーションと合わせて、薬物・疾患に関与する遺伝子群を探索できる。
将来は
- 創薬プロセスの基本ツールとして利用される。
誰が使うのか
- 開発段階ではライフサイエンス関係の研究者及びベンチャー企業。
- 完成時には製薬関連企業及びシステムズバイオロジー研究者。
平成20年度実施計画
1)ネットワーク推定アルゴリズムのペタスケール計算化技術の開発
これまでの大規模遺伝子ネットワークの推定技術では、ネットワークという形で探索できた遺伝子数は約1000が限界であり、これは数としては、ヒト遺伝子数の3%程度である。さらに、生物は、遺伝子ごとの発現制御をしているのではなく、遺伝子に平均五個以上あるプロモーターごとに遺伝子を使い分けているという事実を考えると、少なくとも数万ノードのネットワーク推定とその解析を可能とする技術ができなければネットワークの網羅的な探索により、薬の被影響遺伝子の同定、作用点の推定、副作用の予測・回避、創薬ターゲット・毒性関与パスウェイ探索などを大規模に行うことはできない。本研究項目は、この規模に対応するためのネットワーク推定技術を開発することを目標とする。今年度は、平成19年度までに検討した結果及び研究成果に基づき、中規模(数百~数千)ネットワーク推定方式のプログラム化および有効性の検証実験を行う。具体的には、上位構造に基づく最適なベイジアンネットワークの探索アルゴリズムのプログラム化、グラフィカルガウシアンモデルに基づいた遺伝子ネットワーク方式のプログラム化、状態空間モデルによるモジュールネットワーク推定に基づく遺伝子ネットワーク推定方式のプログラム化を開始し、シミュレーション数値実験による方式の検証、公開・非公開データ及び3)の実験データを用いた方式の改良及び問題点の追及を試みる。併せて、これらのプログラムの改良に必要な要素技術研究を行う。そして、平成20年度から開始する研究項目4)のデータ解析融合プラットフォームに組み入れられるように遺伝子ネットワーク探索ソフトウェアの開発を目指す。
2)データ同化による生命体ネットワーク推定技術の開発
本研究項目では、ペタスケール計算の能力を用いて、生命体のシミュレーションモデルに、データの情報を動的かつ適切に取り込むためのデータ同化技術を開発することにより、モデル及びデータ、それぞれ単独では得られない有用な情報を抽出することが可能なデータ駆動型の大規模データ解析及びシミュレーション技術を開発することを目標とする。今年度は、平成19年度までの検討結果及び研究成果に基づき、データ同化による中規模生命体ネットワーク、パラメータ推定技術の開発、及び一部実データでの検証実験を行う。具体的には、「次世代生命体統合シミュレーションソフトウェアの研究開発」(生命体シミュレーションのためのデータ同化技術の開発・統計数理研究所・樋口知之教授)と共同で研究してきた成果に基づき、その方式のソフトウェア化を検討する。このために、Cell Illustratorを用いてCSMLで記述されたEGFRシグナル伝達・遺伝子発現ネットワーク関する素モデルを利用し、研究項目4)で開発する実験データを用いて、時系列のタンパク質及び遺伝子発現データをCSMLモデルに同化させる計算機実験を行い、技術の評価と改良を行う。そして、統計数理研究所・樋口知之教授のグループと共同して技術開発を行い、研究項目3)のデータ解析融合プラットフォームに組み入れられるように生命体データ同化エンジンの開発を目指す。
3)データ解析融合プラットフォームの開発
本研究項目は、データ解析融合チームで開発した技術とプログラムを、相互に連携させるソフトウェアプラットフォームを構築し、それを活用し、ゲノムから分子のネットワークをつなぐことにより初めて実現可能な情報抽出・予測・発見へ至る道筋を示すことを目標とし、それにより創薬ターゲット探索および個人差を考慮した医療のための基盤情報技術の創出を目指す。そのために、既存の商用・非商用GUIソフトウェア及びデータベースと本チームで開発されるソフトウェアを統合したソフトウェア環境を一つの利用形態事例として構築する。今年度は、Cell Illustratorをモデリング・GUIツールとして利用し、その他にBIOBASE、IPAなどのデータベースを利用し、ネットワーク推定、データ同化、データ解析、シミュレーション、データベースを生命分子のネットワークでつなぐためのプログラム開発に着手する。
4)遺伝子ネットワーク推定及びデータ同化技術開発のための実験データ取得
本研究項目では、遺伝子ネットワーク推定及びデータ同化技術の開発のために、生命体ネットワークのモデル系として、EGFRシグナル伝達・遺伝子発現ネットワークを一般モデルの研究対象とし、正常肺と非小細胞肺癌における必要なデータを取得することを目標とする。このデータを、GefitinibなどのEGFR分子標的薬の効果予測バイオマーカー候補の抽出と、これらの薬剤に対する耐性パスウエイの解明から新たな分子標的候補を抽出するためのネットワーク応用技術につなげる。このため、今年度は、まずヒト初代肺胞上皮細胞内におけるEGFRシグナル伝達・遺伝子発現ネットワークに関するデータを取得する。これに基づいたネットワークを正常肺におけるゴールデンスタンダード(一般モデル)と位置づける。一般モデルに「個」のデータを同化させ、それをバイオマーカー候補の抽出技術へとつなげるために、EGFR分子標的薬Gefitinib感受性のヒト非小細胞肺癌由来細胞株PC9並びに、PC9由来Gefitinib耐性亜株、加えて不死化肺胞上皮由来細胞に野生型EGFRあるいはGefitinib感受性変異EGFRをトランスフェクションにて構成的に発現させた細胞、さらに他の複数のヒト非小細胞肺癌由来細胞株を用いて、これら肺癌細胞内における異常なEGFRシグナル伝達・遺伝子発現ネットワークに関するデータを取得する。具体的には、Gefitinib存在下非存在下において、EGFR活性化によって惹起される、遺伝子発現の動態変化の詳細な時系列遺伝子発現データをアレイ解析により取得する。その中から転写産物量が有意に変動する数百~数千の遺伝子を抽出する。また、100種程度のタンパク質について抗体を用いて定量を行い時系列高精度のプロファイルを取得する。
平成19年度実施計画
1)ネットワーク推定アルゴリズムのペタスケール計算化技術の開発
これまでの大規模遺伝子ネットワークの推定技術では、ネットワークという形で探索できた遺伝子数は約1000が限界であり、これは数としては、ヒト遺伝子数の3%程度である。さらに、生物は、遺伝子ごとの発現制御をしているのではなく、遺伝子に平均5個以上あるプロモーターごとに遺伝子を使い分けているという事実を考えると、少なくとも数万ノードのネットワーク推定とその解析を可能とする技術ができなければネットワークの網羅的な探索により、薬の被影響遺伝子の同定、作用点の推定、副作用の予測・回避、創薬ターゲット・毒性関与パスウェイ探索などが大規模に行うことはできない。本研究項目は、この規模に対応するためのネットワーク推定技術開発を行う。これまで、ベイジアンネットワークをはじめとして、マイクロアレイなどによる遺伝子発現データから遺伝子間の発現因果に関するネットワークを推定するアルゴリズムが研究されてきたが、本年度は、数万遺伝子を対象として、こうしたアルゴリズムをペタスケールで実装しパフォーマンスを出すための実装技術を開発するなかで技術的問題を同定・解決していくとともに、ペタスケール計算が実現されたときに、生命体システムの中のネットワークについて新たにどのような知見がえられる可能性があるかについてそのビジョンを描く。
2)データ同化による生命体ネットワーク推定技術の開発
本研究項目では、ペタスケール計算の能力を用いて、生命体のシミュレーションモデルに、データの情報を動的かつ適切に取り込むためのデータ同化技術を開発することにより、モデル及びデータ、それぞれ単独では得られない有用な情報を抽出することが可能なデータ駆動型の大規模データ解析及びシミュレーション技術を開発することを目標とする。本年度は、比較的小規模(数十変数)のパスウェイモデルを文献及びデータから構築し、それに対して、具体的データを用いて、現段階で、本データ解析融合チームで開発されているデータ同化技術を適用して、生命科学的な視点からデータ同化結果を評価し、その技術的問題及びペタスケール計算化のためのロードマップを描く。
平成19年度実施内容(成果)
1)ネットワーク推定アルゴリズムのペタスケール計算化技術の開発
グラフィカル・ガウシアン・モデルによる大規模遺伝子ネットワークの推定手法を開発した。複雑な変数間の関連構造を探索する手法の一つとして、グラフィカル・ガウシアン・モデリングが提案されている。この手法では、多変量正規分布の仮定の下で、変数間に成り立ついくつかの条件付き独立性を規定したモデル(共分散選択モデル)を推定することにより、変数間に成り立つ独立性・条件付き独立性を無向グラフにより簡潔に表現することができる。近年では、グラフィカル・ガウシアン・モデルは、バイオインフォマティクスの分野において、マイクロアレイデータから遺伝子ネットワークを推定する有用な手法として注目されている。しかしながら、大規模遺伝子ネットワークの推定については、従来法による推定では満足な推定精度は得られておらず、特に数万から数十万遺伝子を対象とした遺伝子ネットワークの推定では、モデルのパラメータ推定自体が困難なのが現状である。グラフィカル・ガウシアン・モデルのパラメータ推定において本質的な問題は、分散共分散行列の逆行列の推定である。実際のモデリングでは、観測された標本分散共分散行列の逆行列を計算した上でいくつかの要素を0 とおいた相関構造モデルにより、観測された標本分散共分散行列を近似する過程を繰り返すことにより、無向グラフを推定する。しかしながら、マイクロアレイデータなどの超高次元データでは、標本数が変数よりも少ない場合が多く、この場合、標本分散共分散行列の逆行列を直接計算することができないため、グラフを推定することが困難となる。また、どの分散共分散行列の要素を0 とおくかはデータから判断しなければならず、変数が10000 個のとき、全部で210000×9999/2 通りという多数のモデルが存在する。したがって、これらのモデルの中から一つモデルを選択するルール、およびそのモデルが適切なモデルかどうかを判断する基準が必要となる。以上の問題点に対し、本研究課題では、共分散選択モデルを線形回帰モデルの変数選択の枠組みで捉え、そのモデルのパラメータ推定をL1 正則化法によって推定する手法を提案した。提案する手法の利点の一つは、標本数が変数よりも少ない場合でもグラフを推定できる点が挙げられる。また、正則化項としてパラメータに対してL1 ノルムの制約を用いているため、パラメータを推定すると同時にパラメータに含まれるいくつかの要素が0 になり、これによりグラフの構造が自動的に選択される。加えて、従来用いられていたL1 ノルムの等方的制約をデータからより柔軟な非等方的制約に改良することにより、擬陽性の辺を削減し、ネットワークの推定精度を大幅に改善する手法(Weighted Lasso)を提案した。これらの提案する推定法によるグラフィカル・ガウシアン・モデルの構築に当たっては、L1 正則化法における正則化パラメータの選択が本質的な問題となる。この選択問題をベイズ推論の観点から考察し、適切なモデルを選択するための一つのモデル評価規準(NEBC)を導出した。さらに、シミュレーション、および実データの解析により、提案手法は従来法よりもネットワークの推定精度に関して優れていることを示した。当該年度の新規推定手法の開発により、従来法では困難であった数万の大規模遺伝子ネットワークの推定が可能となった。具体的には、標本数400 の遺伝子破壊株のマイクロアレイデータから遺伝子数13732 の大規模遺伝子ネットワークを推定することに成功した。もう一つの成果として、ベイジアンネットワークにより大規模遺伝子ネットワークを推定するためのアルゴリズムを開発した。確率的グラフィカルモデルの一つであるベイジアンネットワークは、マイクロアレイデータに基づく遺伝子ネットワーク推定において極めて広く用いられている。遺伝子ネットワーク推定においては、遺伝子はグラフ上のノードとして表現され、それらの依存関係は、ノードとノードを結ぶエッジによって表現される。このとき、遺伝子ネットワークの推定とは、計測データに基づくグラフィカルモデルの構造学習に他ならない。しかしながら、ベイジアンネットワークの構造探索は、NP 困難であることが示されている。特に、多くの遺伝子を含むネットワークの構造学習に対しては、発見的アルゴリズムを使用せざるを得ず、その精度低下に大きな課題を残している。ベイジアンネットワーク構造探索のためのアルゴリズムは、独立性検定に基づく方法と、スコア関数を用いた方法の二種に大別される。独立性検定は、ベイジアンネットワークにおいて本質的な条件付き独立性を直接評価するため、小規模なネットワークの構築においては有効である。しかしながら、大規模なネットワークを推定するためには、統計的仮説検定を膨大な数繰り返す必要があり、多重検定の補正は大きな問題となる。また、データのノイズに敏感であり、構造推定の誤りが累積し結果として得られるネットワークの精度は低下する。一方、スコア関数を用いる方法は、探索するグラフの空間はsuper exponential の大きさであり、最適解探索は不可能となる。従って、greedy 探索による局所最適解を得るにとどまっている。近年、これら二種の方法を融合したMMHC は、精度と計算速度の両面において優れていることが経験的に示されている (Tsamardinos et al. 2006)。MMHC は、まず、独立性検定の方法であるMMPC を用いて真のグラフのskeleton を近似する。次に、学習したskeleton に制限した探索空間において、スコア関数を利用したgreedy 探索を行う手順となっている。本研究課題において、新しい構造制約として無向グラフにより表される super-structure を提案した。このとき、我々の問題は、「与えられたsuper-structure S にskeleton が含まれるような非閉路有向グラフの空間におけるスコア関数の最大化」と定義される。Super-structure による制約は、MMHC における制約と比較すると、skeletonを部分的にしか制限せず、より柔軟なものである。また、真のsuper-structure は、真のネットワークのskeleton よりも極めて容易に近似できることから、より現実的であると考えられる。加えて、我々は、より大きな有意水準を用いたMMPC によりsuper-structure を学習する方法を提案する。統計的仮説検定における多重性の補正としては、ボンフェロニの補正が頻繁に用いられる。しかしながら、ボンフェロニの補正は、有意と判定した仮説に一つの擬陽性も許さない極めて厳しいものである。緩いが現実に即した制約として提案したsuper-structure の構成にはこのような厳しい補正は適切ではない。また、数百万回と繰り返される仮説検定における有意水準の補正を厳密に行うのは現実的ではない。一方、super-structure は、真の構造のskeleton を含んでいればよいので、かなり緩い有意水準での評価が許される。この点もMMHC と比較した際の提案法の利点である。このsuper-structure を用いて、Ott et al. (2004) の提案したOS (Optimal Search) アルゴリズムをsuper-structure に制限することで拡張し、COS (Constraint Optimal Search) アルゴリズムを提案した。つまり、super-structure が与えられたもとで、COS アルゴリズムは最適なネットワーク構造を求めることができる。COS アルゴリズムの計算科学的複雑度は、S の連結部分集合 Cons(S) の数に依存する。そこで、Cons(S) を厳格に表すデータ構造を開発し、COS アルゴリズムの効率的実装を行った。また、アルゴリズムの上限・平均複雑度を導出し、それらがS の平均次数に依存することを示した。この結果は、super-structureが疎な場合には、COS アルゴリズムは計算量、メモリー使用量を大幅に削減できるため、OSアルゴリズムより大きなネットワーク推定に適用できることを示している。 Ott et al.(2004) の方法では、200G フロップスのスーパーコンピュータSun Fire 15K を利用しても高々30 遺伝子程度のネットワークの完全探索しかできなかった。しかしながら、super-structure の平均次数を遺伝子ネットワーク推定において現実的である2 程度に設定したとき、2 倍以上、具体的には60 から80 程度の遺伝子を含むネットワークの(super-structure という制約付き) 完全探索が可能となった。また、super-structure をデータから近似する現実的状況においても、このアルゴリズムにより推定されるネットワークは、 greedy 探索やMMHC アルゴリズムによるものよりも精度が高いことを大規模な数値実験を通して示した。以上、これらの2つの成果により、ペタスケール計算で、遺伝子ネットワーク推定の大規模化・精緻化を可能とする目処がたった。これらのアルゴリズム群はプログラム化し、「遺伝子ネットワーク探索ソフトウェア(仮称)」に統合中である。
2)データ同化による生命体ネットワーク推定技術の開発
当該年度は、生命体データ同化の実現に向けた必須要素技術の開発を、統計数理研究所樋口グループと共同して行った。生命体データ同化では、パスウェイシミュレーションモデルと観測データ(遺伝子発現、タンパク濃度時系列データ等)の情報を、合理的に統合することにより、数値シミュレーションおよび観測データ、双方単独では表現できない情報を抽出することを狙いとする。それにより非観測要素(観測されていないがシミュレーションモデルには含まれるタンパクの濃度変化、反応速度係数等)の予測や推定、また複数のシミュレーションモデル群の生成・評価を通じた、生体制御ネットワークに関する新規仮説群の生成・評価および、より現実に即したモデルへの改良等が期待される。上述の生命体データ同化を実現するためには、さまざまな要素技術の開発が必要となる。当該年度は、特にパスウェイシミュレーションモデルのパラメータ推定手法、モデル選択手法について開発を行った。開発に当たっては、開発用の試験系として、動的システムを記述する視覚的プログラミング言語Hybrid Functional Petri Net (HFPN)により作成された、パラメータが数十程度の比較的小規模な、マウス概日周期パスウェイシミュレーションモデルを用いた(Fujii et al. 2004)。データ同化の問題は、非線形状態空間モデルにより定式化されることから、それぞれの技術開発における問題は、非線形状態空間モデルにおける、パラメータ推定問題、モデル選択の問題に、それぞれ帰着される。前者のパラメータ推定の問題に関しては、最適化に基づく手法、およびマルコフ連鎖モンテカルロ(MCMC)法に基づく手法の開発を行った。具体的には、最適化に基づく手法では、HFPN で使われる反応のクラスをうまく制限することにより、目的関数をsemi-convex optimization を用いて高速に推定する手法の開発を行った。MCMC に基づく手法では、状態空間モデルの確率モデルの構造を用いて、パラメータ変数および状態変数の実現値をGibbs 法とMetropolis-Hastings法を組み合わせたサンプリングにより発生させ、推定値を得る手法の開発を行った。結果、生命体データを用いたデータ同化問題の特徴として、少数時点の時系列観測データ点から、多数のパラメータを推定する必要があり、推定には困難さが伴うが、ベイズ統計の枠組みによりパラメータに事前分布を投入することで、シミュレーションデータを用いて数十程度のパラメータの同時推定が可能となった。後者のモデル選択に問題に関しては、最適化手法により推定されたパラメータを用いて、異なる構造をもつパスウェイシミュレーション間の良さを比較するための、ベイズ型情報量規準を導出した。更に、Ueda et al. (2002)に掲載されたマウス概日周期遺伝子発現時系列データに対して、上記のモデルと、最適化手法によるパラメータ推定手法を適用することにより、非観測タンパク質群の時系列変動を推定し、また情報量規準を用いたモデル比較手法により、新規仮説パスウェイモデルを生成した。これらの技術は、プログラム群として「生命体データ同化エンジン」に統合する予定である。また次年度以降の技術開発の問題として、生命体データ同化技術が、これまで試みられていない新たな技術であり、もともとデータ同化を想定して取得された観測データはもとより、既存の観測データのうちデータ同化に利用できるものが、まだほとんど存在しないことが挙がっていた。そこで、次年度以降の開発を見据え、当該年度は開発用のテスト系として、EGF シグナル伝達系のパスウェイを選び、モデル構築および、データ同化に適したデータ計測計画を作成した。モデル構築においては、選定した系に対する中規模(数百変数)の初期パスウェイモデルを文献から、HFPN を用いてCell Illustrator 上で構築した。データ同化用のデータ取得については、平成20 年度に平成19 年度に得られた知見に基づいて行う。